-
Deep learning based drug-protein interaction (1)AI/Bio & Medical 2022. 12. 26. 17:48
Naver D2 강의를 듣고 정리한 내용입니다.
강의 주소 : https://www.youtube.com/@naverd2848
naver d2
NAVER D2(https://d2.naver.com)의 공식 YOUTUBE 채널입니다.
www.youtube.com
1. Probelm definition
1.1 Drug discovery process

Drug discvoery의 pipeline은 크게 위와 같이 두 가지로 나눌 수 있다.
위 그림에서의 색깔 영역(초록과 노란색 영역)이 의미하는 다음과 같다.
초록색 : Physical(실험관) or computer based(in-silico) experience
노란색 : 동물이나 사람에게 실험 (임상 실험)
초록색에 해당하는 실험관과 컴퓨팅 기반의 실험으로 실험해볼만한 약물의 list를 좀 줄이고 노란색 부분이에서 동물이나 사람에게 실험을 진행하여 FDA의 승인을 받는 과정이라고 정리할 수 있다.
일반적인 스타트업 회사에서는 노란색 부분의 과정은 너무 많은 cost를 필요로하여 진행하지 못하고 대부분이 초록색 부분의 과정에 초점을 맞추어 진행한다. 이때, 초록색 부분 또한 아래와 같이 3개의 sub-task로 나눌 수 있다.
Target Indentification :
대부분의 질병은 단백질의 이상 발현으로 생긴다. 어떤 질병이 어떤 단백질의 이상 발현으로는 질병이 생긴것 인가? 이것을 target이라 하는데 이 target를 찾는 과정이다.
Molecule Discovery :
target을 조절할 수 있는 약물(molecule)을 찾는 과정이다.
Molecule Optimization :
모든 약물은 간에서 대사가 일어나서 독성이 생기며 molecule이 크면 합성하는데 돈이 많이 들고 어려운 특징이 있다. 그런 molecule property들을 최적화 시키는 과정이다. 그냥 나온 molecule을 바로 사용하는 것이 아닌 molecule의 독성을 낮추고 weight를 줄이는 과정은 반드시 필요하다.
이 세 가지 과정 중에 Molecule Discovery에서 두 가지 과정이 존재한다. 그것은 Drug Repurposing과 Generating이다.
Drup Repurposing은 기존에 있던 약을 새로운 용도로 사용하는 것이며 Drug Generating은 그 target을 효과적으로 control 할 수 있는 이전에 없던 약물을 만드는 것이다.
1.2 Drug Repurposing

위 표를 보면 Antihitamine(항히스타민)으로 사용되는 Astemizole이라는 약이 있다. 근데 실험을 해보는 Malaria에 이 약이 효과 있어다고 표에 나와 있다. 이렇듯 기존의 약물을 다른 질병 치료를 위해서 사용되는 것이 Drug Repurposing이다.
표의 왼쪽에 있는 약물 들은 임상 실험이 다 끝난 즉, 독성 test등이 끝나 사용이 가능하다고 승인을 받은 물질이기 때문에 안전하다는 것을 의미한다. 그렇기에 Drug Repurposing을 하면 임상 1상(※ 임상 1상은 독성 test를 하는 단계이다 ※)을 건너뛸 수 있다는 장점이 있다. 임사하는 것에 시간과 돈이 매우 많이 드는데 이것을 절약할 수 있다는 것은 매우 큰 장점이다. 그렇기 때문에 많은 스타트업 기업에서 시도를 하고 있다.
2. Background
2.1 Drug Target Interaction
Drug Target Interaction 문제를 정리하면 다음과 같이 나타낼 수 있다.
Input : [ Drug - molecule ], [ Target - protein(biomarker) ]
Output : Interaction (affinity score)
Example : EGFR protein (cancer biomarker) has high affinity score with Laptinib (anti-cancer durg)
If other non anti-cancer drug has high affinity socre with EGFR, they can be candidates of an anti-cancer durg
위 과정을 설명하면 input으로 drug와 protein이 들어가고 output은 이 둘 사이의 얼마나 binding이 일어나는지를 숫자인 regression model을 만드는 것이 목표이다.
예를 들어, 대부분의 암에서는 EGFR이라는 단백질의 이상 발현이 굉장히 많이 관측 된다. 그 단백질과 최근에 많이 쓰이는 항암제 주엥 하나인 Laptinib을 사용하면 affinity score가 굉장히 높게 올라가게 된다. 그럼 만약 항암제가 아닌데 EGFR과 잘 붙는 약이 있다면? 우리는 그 약을 항암제로 test해볼만 하다라고 볼 수 있는 것이다. 이것이 drug repurposing이다.
2.1.1 Inputs of DTI
Molecule (SMILES format)
Laptinib : "CS(=O)(=O)CCNCC1=CC=C(O1)C2..."
Protein (FASTA fromat)
EGFR : "MRPSGTAGAALLALLAALCPASRALE..."
위는 DTI의 input을 정리해 본 것이다.
Laptinib -> SMILES 형태로 Atom의 조합이다.
EGFR -> FASTA 형태로 제공되며 표시된 영어는 전부 아미노산이다. 즉, 아미노산 서열로 표기가 된다.
결론적으로 molecule과 protein은 전부 sequence data이다.
2.2 Sequence Representation
Sequence : SMILES, FASTA, and test
< Vector representation >
One hot vector
(word/character) Embedding - more information
Once represented as a vector, we can apply many deep learning methods
위에서 설명된 것과 같이 sequence를 먼저 vector형태로 representation 해주어야 deep learning에 적용할 수 있다. vectorization해주게 되면 어떠한 deep learning model에도 사용이 가능해진다. 이때 One-hot vector 혹은 Embedding 기법을 사용하게 되는데 embedding 기법을 사용할 시에 one-hot보다 더 많은 정보를 사용할 수 있게 된다. 그러한 이유로 embedding에 관한 내용을 더 자세히 살퍄볼 것이다.
Word embeddings로는 다음과 같은 방법들이 있다.
- Local contextual embeddings : Word2Vec
- RNN based contextual embedding : ELMO
- Attention (w/o RNN) based contextual embedding : Transformer
- BERT (Transformer + Masked LM)
2.2.1 Word2Vec
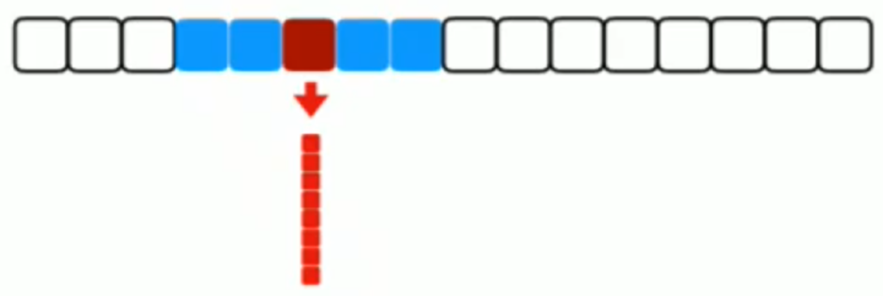
Word representation : wrod -> vector
W2V : local context words when calculation a representation vector for a target word
ex) When inferring the red word, 4 context words (blues) are used
파란색 token의 정보들을 이용하는 것 -> local context를 보는 것
# How to train word2vec?
Example sentence : I go to Emory University located in Atlanta.
input – context words : “I”, “go”, “Emory”, “University”
output – target word, “to”
우리가 위 문장에서 "to"라는 단어를 represents하려면 앞뒤로 2개의 단으로 떼어내어 위에 표시된 단어 4를 input으로 사용한다.
하나의 단어당 하나의 vector가 나온다는 단점이 존재한다. 예를 들어, apple의 경우 회사일수도 있고 과일일 수도 있는데 하나의 vector만 나온다는 문제점이다.
2.2.2 ELMO

Concates of independently trained left-to-right and right-to-left LSTM
It considers all words in a sentence to represent a word
Long sequence -> information vanishing problem
W2V의 단점을 개선하고자 context를 전부 반영해보라!라는 아이디어로 나온 것이 ELMO이다.
그림의 빨간색 단어를 얻기 위해 좌측 부분을 RNN으로 전부 훑어본 다음 붙이고 우측도 RNN을 사용하여 vector를 만들어 그 두 vector를 concatenate하여 단어를 얻는 방법으로 이렇게 진행하면 문장에 따라서 이 target vector가 계속 바뀔 것이다.
# How to train ELMO?
For the simplicity I assume word level embeddings (but actually they use character level embeddings)
Example Sentence : I go to Emory University located in Atlanta.
Input :
left-to-right model : “I“, “go”
right-to-left model : “Atlanta”, “in”, “located”, “University”, “Emory”
Output : “to”
EMLO의 가장 큰 문제점은 LSTM과 같은 RNN의 단점을 그대로 이어 받았다는 것이다.
우측부터 쭉 학습을 한다고 가정하면 우측부터 있는 정보는 희석이 된다. 만약 그림의 빨간색 단어를 추출하기 위한 가장 중요한 정보가 우측 끝에 있다고 가정을하면 상당히 큰 손실이 발생하는 것이다.
즉, Long Sequence에서 information vanishing 이 생기는 문제가 있다.
2.2.3 Transformer

Calculating a vector for a word using all words in the sentence
Attention is all you need 논문을 통해서 self-attention이 제안되었다.
그에따라 Embedding + RNN를 Transformer(self-attention)로 변경하였다.
빨간색 token을 represents 하기 위해서 이 token을 포함하고 있는 모든 token을 weight sum해버리는 방식이다.
여기서 중요한 점은 self-attention layer를 Embedding layer와 RNN layer를 모두 대신 한다는 특징을 가지고 있다.
Transformer의 구성에 대해서 그럼 자세하게 알아보자.
2.2.3.1 Encoder-Dencoder in Transformer

Transformer는 encoder라는 block들이 쌓여서 최종 encoding을 진행하고 그 정보를 바탕으로 decoding을 쌓아서 output을 얻는 구조이다.
encoder block은 크게 Self-Attention과 Feed Forward block으로 나누어져 있다.
# Encoder

input에 두 개의 token이 들어가면 random vector로 표현이 되고 self-attention을 거치면 z라는 hidden vector로 표현이 되게 된다. z가 feed forward layer를 거쳐서 최종 하나의 encoder output인 r로 표현이 된다.
# Self-Attention High level explanation

it_ 이라는 toke을 포현하기 위해서 it을 포함한 모든 token들의 weight들을 모두 계산한 다음 그 token들의 vector들을 저 weight를 이용해서 weight sum한다.
# Weighte Sum
Weighted sum을 하려면 여러가지 helper vector가 필요한데 이 transformer에서는 3가지 helper vector를 사용한다.
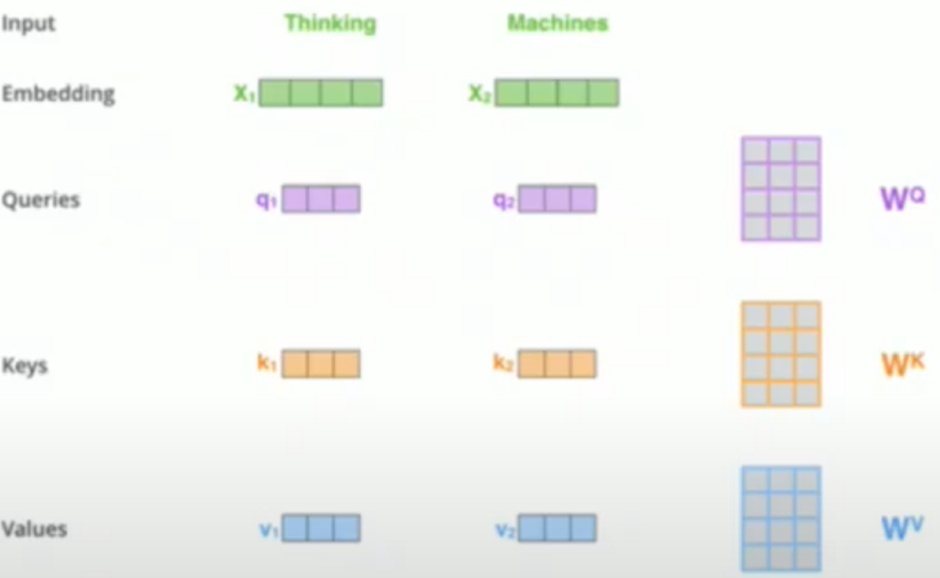
Query, Key, and Value vector are used when calculating hidden representations
These helper vectors are just a projection from trainable params Wq, Wk, and Wv
초록색 embedding vector에서 단순히 W^q, W^k, and W^v 라는 parameter에다가 그냥 projection 한 결과이다.
# Scoring

Calculate scores for each word with respect to token “Thinking” using (query, key)
For example : “Thinking” -> 112, “Machines” -> 96
Repeat this for all other tokens
The vector, values, will be used in the next step
Weighted sum을 먼저 계산하기 전에 각각 word score를 구하는 것이 필요하다.
그러기 위해서는 각각의 vector가 무엇을 의미하는지 알아야한다.
query vector : 어떤 단어를 기준으로 score를 계산하는지 (주인)
key vector : 그 단어가 어떤 token을 대상으로 weight를 구하고 있는지 (target)
value vector : 저 embedding을 weighted sum 할때 한번 더 projection 결과를 weighted sum 하는 vector
Thinking token 관점에서 score를 계산해 볼 때 query 가 주인이고, thinking에서 보는 thinking의 token의 weight을 구하기 위해서 q1 * k1, thinking 관점에서 machines token의 weight를 구하기 위해서는 q1 * k2 이다.
# Self-Attention

Divide by 8
The square root of the dimension of the key vectors (paper used dim=64)
Softmax : Normalized scores to be sum to one
Hidden representation is a weighted sum of value vectors
weight 구하기 전에 122 -> 14 -> 0.88 로 바뀌게 되는데122->14가 되는 과정은 122를 8로 나누어 준 것이다.
이때 8은 magic number가 아니라 numerical이 좀 stable하게 해주는 divider 이다. 이때 이수는 dimension을 루트 씌워준 값으로 구하게 된다.
이렇게 구해진 8을 122에 나누게 되면 14가 나오게 되고 이것을 softmax에 넣으면 0.88을 얻게 된다.
# Multi-Heads
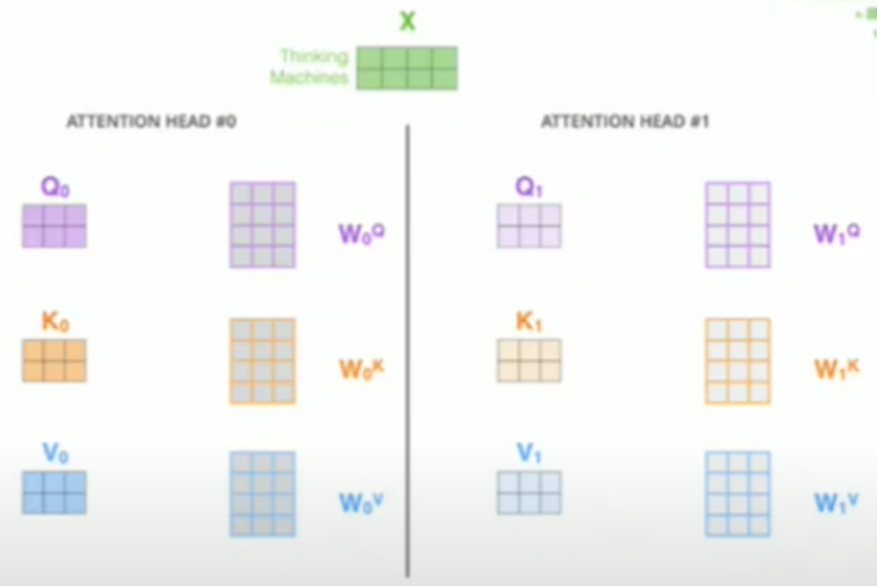
Multi filters in CNN, Multi heads in Transformer
8 Heads : 8 sets of trainable params Wq, Wk and Wv, 8 sets of z1 and z2
Expecting different heads to learn different aspects
일반 CNN에서 filter를 여러 번 적용하게 되는데 이것과 같다고 볼 수 있다.
self-attention 에서는 q, k and v를 다른 initialization으로 여러 번 반복하게 되는데 이를 Head라고 부르는 것이다.
# FeedForward

multi-head attention (head = 8) 을 거쳐서 나온 8개의 z를 concatenate 하여 커다란 dense network를 지나게 하여 최종 output vector인 R vector를 얻게 된다.
# Positional Encoding

Why PE? – Need to distinguish “ I am a student ” vs “ am student I a “
Special patterns representing order of words
self-attention mechanism 은 weight sum이다. 이때 weight sum은 위의 예시 문장의 차이가 없게 된다. 즉, 순서 정보가 전부 mix되버린다. 그래서 순서 정보가 없기 때문에 embedding level에서 순서 정보를 심어줄 필요가 있다.
너무 내용이 많아 다음 포스팅으로 이어질 예정입니다.
'AI > Bio & Medical' 카테고리의 다른 글